[Part.1] Image Classification on MLOps
이번 포스팅부터 몇차례에 걸쳐 Image Classification을 Kubernetes 환경에서 MLOps tool을 통해 전체적인 파이프라인을 구축하는 작은 프로젝트를 작성하려고 한다. 프로젝트에서 활용한 쿠버네티스 환경 및 Prerequisite는 아래와 같다. (쿠버네티스 환경 세팅 및 설치 방법은 여기를 참고)
Kubernetes
Cluster
- OS: Ubuntu 18.04.6 LTS
- GPU: Tesla V100 x 4
- Docker v20.10.8
- Kubeadm(on-premise) v1.21.7
- kubelet,kubectl v1.21.7
Client
- OS: mac(Intel)
- Docker v20.10.8
- kubectl v1.21.7
- kubeflow: v1.4.0
- helm v3.7.1
- kustomize v3.10.0
Dataset
https://www.kaggle.com/arunrk7/surface-crack-detection
- 금이간 콘크리트 표면 이미지(Positive) 2만장 + 정상 이미지(Negative) 2만장 = 4만장의 Image Dataset
Content
1. Define Persistent Volume Claim
2. Kubeflow Pipeline
3. Preprocessing
4. Hyperparameter Tuning (weights and biases)
5. Model Training & Test (kfp)
6. Model Versioning (mlflow)
7. Model Serving (bentoml)
8. Monitoring (prometheus & grafana)
1. Define Persistent Volume Claim
Pod 내부에서 작성한 데이터는 기본적으로 언제든지 사라질 수 있기에, 보존하고 싶은 데이터가 있다면 Pod 에 PVC 를 mount 해서 사용해야 한다.
현재 상황의 경우 모델 학습을 위한 데이터가 항상 보존되어야 하기 때문에 PV, PVC를 먼저 생성한다. PV와 PVC는 manifest(yaml 파일)를 직접 작성하여 생성할 수 있지만 kubeflow dashboard에서도 쉽게 생성할 수 있다.
Dashboard의 메뉴에서 Volumes로 PV만 따로 생성할 수 있지만 아래 그림처럼 Notebooks에서 우측 상단 NEW NOTEBOOK을 통해 진행하면 PVC에 대한 스펙을 직접 정의할 수 있으며 주피터랩으로 PV에 직접 접근할 수 있다.
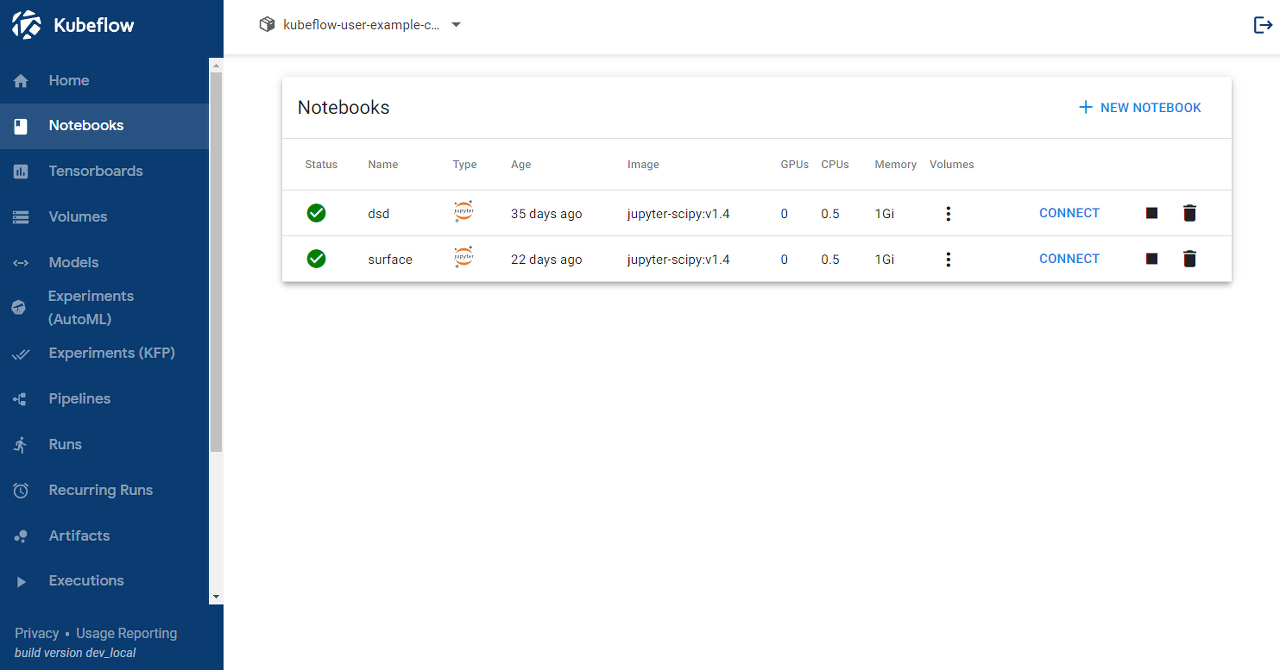
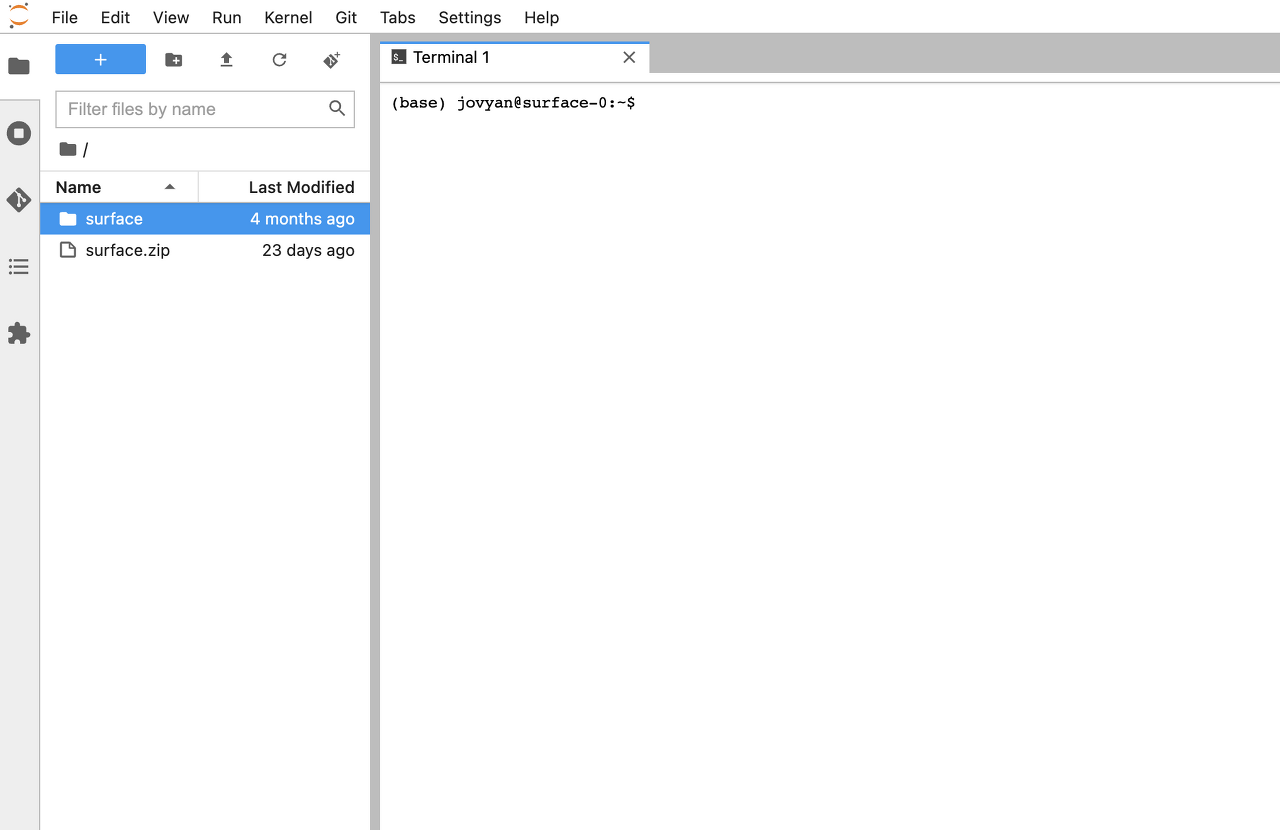
workspace-surface라는 이름의 PVC를 생성하였고 CONNECT에 진입하여 아래와 같이 데이터를 업로드하였다.
2. Kubeflow Pipeline
Kubeflow에서는 Python rapper를 통해 Pipeline을 구축하여 실행하면 자동으로 manifest 파일을 작성해준다. Pipeline은 Anaconda 가상환경에서 진행하였고 아래 라이브러리를 설치하였다.
- Python 3.7
- kfp 1.8.11
- pytorch 1.10.2
- timm 0.5.4
- wandb 0.12.10
- albumentations 1.1.0
- split-folders 0.5.1
- mlflow 1.23.1
- bentoml 0.13.1
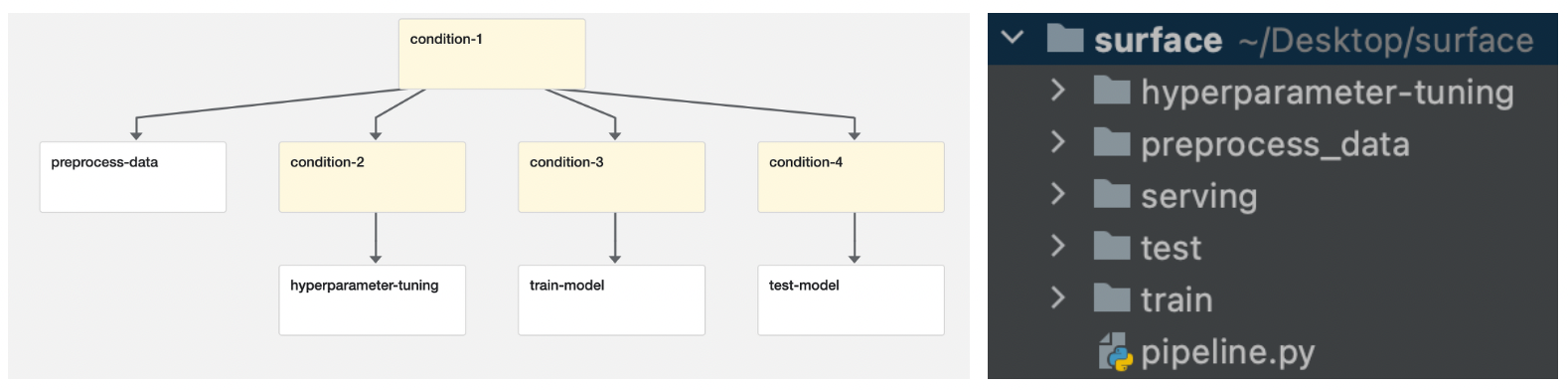
pipeline은 크게 데이터 전처리를 위한 preprocess-data component, hyperparameter-tuning component, 모델을 학습시키고 best모델을 mlflow experiment에 저장하는 train-model component, 저장된 모델을 불러와서 모델의 성능을 평가하는 test-model component 총 4개의 컴포넌트로 구성하였다.
kubeflow pipeline을 작성하는 방법에는 크게 두가지 방법이 있다. 먼저 경량화 컴포넌트를 작성하는방법이 있는데 이 방법은 create_component_from_func라는 kfp의 모듈을 사용해 하나의 함수가 하나의 컴포넌트가 되는 방식으로 하나의 파이썬 파일만으로 모든 컴포넌트를 작성할 수 있어 수정이 빠르고 작성이 간편하다는 장점이 있지만 딥러닝과 같이 코드가 많은 경우에는 오히려 가독성이 떨어지게 된다. 또한 하나의 함수가 개별적인 컴포넌트이기 때문에 컴포넌트에 사용되는 라이브러리를 함수안에서 설치할 버전을 명시하고 import를 해주어야 한다. (+ 파이썬 파일에 한글이 있으면 yaml파일로 작성이 되지 않는다는 단점도 있다.) 아직까지는 불편한 요소가 많기 때문에 경량화 컴포넌트는 가벼운 머신러닝에서 활용할 가치가 있는 것 같다.
또 한가지 방법은 컴포넌트별로 일반적인 딥러닝 파이썬코드를 작성하고 도커 이미지로 빌드 한 후 간단하게 파이프라인에서 연결만 시켜주는 방법이다. 수정할때마다 이미지를 빌드해야 하는 단점이 있지만 한번 잘 구축해 놓으면 가독성도 좋고 관리하기 훨씬 편한 방법인 것 같아 이 방식으로 진행하였다.
먼저 전체 코드를 작성하고 컴포넌트를 하나씩 실행해보면서 어떻게 작동이 되는지 살펴보자
component1: preprocess-data
preprocess.py
import argparse
import os
import numpy as np
import splitfolders
import torch
from torchvision import datasets
from torch.utils.data import DataLoader
import albumentations as A
from albumentations.pytorch import ToTensorV2
def split_dataset(data_path):
splitfolders.ratio(f'{data_path}/surface', output=f'{data_path}/dataset', seed=42, ratio=(0.7, 0.15, 0.15))
# print data amount
data = ['train','val','test']
label = ['negative','positive']
for i in data:
for j in label:
count = len(os.listdir(f'{data_path}/dataset/{i}/{j}'))
print(f'Crack | {i} | {j} : {count}')
def get_mean_std(data_path, img_size):
class Transforms:
def __init__(self, transforms: A.Compose):
self.transforms = transforms
def __call__(self, img, *args, **kwargs):
return self.transforms(image=np.array(img))['image']
dataset = datasets.ImageFolder(f'{data_path}/surface', transform=Transforms(transforms=A.Compose([A.Resize(img_size , img_size), ToTensorV2()])))
loader = DataLoader(dataset, batch_size=10, num_workers=0, shuffle=False)
mean = 0.0
for images, _ in loader:
images = images / 255
batch_samples = images.size(0)
images = images.view(batch_samples, images.size(1), -1)
mean += images.mean(2).sum(0)
mean = mean / len(loader.dataset)
var = 0.0
for images, _ in loader:
images = images / 255
batch_samples = images.size(0)
images = images.view(batch_samples, images.size(1), -1)
var += ((images - mean.unsqueeze(1))**2).sum([0,2])
std = torch.sqrt(var / (len(loader.dataset)*img_size*img_size))
mean = list(map(lambda x: str(x) + '\n', mean.tolist()))
std = list(map(lambda x: str(x) + '\n', std.tolist()))
with open(f'{data_path}/mean-std.txt', 'w') as f:
f.writelines(mean)
f.writelines(std)
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--data-path', type=str, help='dataset root path')
parser.add_argument('--img-size', type=int, help='resize img size')
opt = parser.parse_args()
split_dataset(opt.data_path)
get_mean_std(opt.data_path, opt.img_size)
- 데이터셋이 라벨별로 폴더가 나뉘어 있다면 splitfolders라는 라이브러리로 train, val, test set을 쉽게 나눌 수 있다.
- get_mean_std 함수를 통해 데이터셋의 평균과 표준편차를 계산해 txt파일로 저장한다. (학습 및 평가에 사용될 값)
Dockerfile
FROM pytorch/pytorch:latest
RUN pip install -U numpy split-folders albumentations
RUN mkdir -p /app
ARG DISABLE_CACHE
ADD preprocess.py /app/
WORKDIR /app
ENTRYPOINT [ "python", "preprocess.py" ]
component2: hyperparameter-tuning
kubeflow에서는 hyperprameter tuning을 지원하는 Katib라는 컴포넌트가 있지만 UI가 너무 딱딱하고 yaml파일을 작성해야 한다는 불편함이 있어서 weights and biases(이하 wandb)를 활용하였다.
hyperparameter-wandb.py
import argparse
import os
from tqdm import tqdm
import torch
import torch.nn as nn
import timm
from torch.optim.lr_scheduler import CosineAnnealingLR
from util import seed_everything, MetricMonitor, build_dataset, build_optimizer
import wandb
class ConvNeXt(nn.Module):
def __init__(self, num_classes, pretrained=True):
super(ConvNeXt, self).__init__()
self.model = timm.create_model('convnext_tiny', pretrained=pretrained) # timm 라이브러리에서 pretrained model 가져옴
self.model.head.fc = nn.Linear(self.model.head.fc.in_features, num_classes, bias=True)
def forward(self, x):
return self.model(x)
def train_epoch(train_loader, epoch, model, optimizer, criterion, device):
metric_monitor = MetricMonitor()
model.train()
stream = tqdm(train_loader)
for i, (images, targets) in enumerate(stream, start=1):
images, targets = images.float().to(device), targets.to(device)
output = model(images)
loss = criterion(output, targets)
optimizer.zero_grad()
loss.backward()
optimizer.step()
predicted = torch.argmax(output, dim=1)
accuracy = round((targets == predicted).sum().item() / targets.shape[0] * 100, 2)
metric_monitor.update('Loss', loss.item())
metric_monitor.update('accuracy', accuracy)
stream.set_description(
f"Epoch: {epoch}. Train. {metric_monitor}"
)
wandb.log({"Train Epoch": epoch, "Train loss": loss.item(), "Train accuracy": accuracy})
def val_epoch(val_loader, epoch, model, criterion, device):
metric_monitor = MetricMonitor()
model.eval()
stream = tqdm(val_loader)
val_loss = 0
with torch.no_grad():
for i, (images, targets) in enumerate(stream, start=1):
images, targets = images.float().to(device), targets.to(device)
output = model(images)
loss = criterion(output, targets)
val_loss += loss
predicted = torch.argmax(output, dim=1)
accuracy = round((targets == predicted).sum().item() / targets.shape[0] * 100, 2)
metric_monitor.update('Loss', loss.item())
metric_monitor.update('accuracy', accuracy)
stream.set_description(
f"Epoch: {epoch}. Validation. {metric_monitor}"
)
wandb.log({"Validation Epoch":epoch, "Validation loss": loss.item(), "Validation accuracy": accuracy})
wandb.log({"VAL EPOCH LOSS": val_loss / len(val_loader.dataset)})
return accuracy
def main(hyperparameters=None):
wandb.init(project='surface-classification', config=hyperparameters)
config = wandb.config
epochs = 1
# read mean std values
with open(f'{opt.data_path}/mean-std.txt', 'r') as f:
cc = f.readlines()
mean_std = list(map(lambda x: x.strip('\n'), cc))
model = ConvNeXt(num_classes=2, pretrained=True)
model.to(device)
train_loader, val_loader, _ = build_dataset(opt.data_path, config.img_size, config.batch_size, mean_std)
optimizer = build_optimizer(model, config.optimizer, config.lr)
criterion = nn.CrossEntropyLoss()
scheduler = CosineAnnealingLR(optimizer, T_max=10,
eta_min=1e-6,
last_epoch=-1)
for epoch in range(1, epochs + 1):
train_epoch(train_loader, epoch, model, optimizer, criterion, device)
val_epoch(val_loader, epoch, model, criterion, device)
scheduler.step()
def configure():
sweep_config = \
{'method': 'random',
'metric': {'goal': 'minimize', 'name': 'VAL EPOCH LOSS'},
'parameters': {'batch_size': {'values': [32, 64, 128]},
'epochs': {'value': 1},
'img_size': {'values': [112, 224]},
'lr': {'distribution': 'uniform',
'max': 0.1,
'min': 0.001},
'optimizer': {'values': ['adam', 'sgd']}}}
return sweep_config
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--data-path', type=str, help='dataset root path')
parser.add_argument('--device', type=str, help='cuda device, i.e. 0 or 0,1,2,3 or cpu')
opt = parser.parse_args()
if opt.device == 'cpu':
device = 'cpu'
else:
os.environ["CUDA_VISIBLE_DEVICES"] = opt.device
device = "cuda" if torch.cuda.is_available() else "cpu"
print(f"DEVICE is {device}")
seed_everything()
wandb.login(key='write private key')
hyperparameters = configure()
sweep_id = wandb.sweep(hyperparameters, project='surface-classification')
wandb.agent(sweep_id, main, count=10) # count: 실험 횟수
- 모델은 timm 라이브러리에서 pretrained ConvNeXt-tiny를 사용
- 하이퍼 파라미터 튜닝은 random search 방식으로 최적의 batch_size, img_size, learning rate, optimzer를 찾는 것이 목표
- wandb.login에서 자신의 wandb 계정 private key를 붙여넣어 계정을 연동해야 한다.
util.py
import os
from collections import defaultdict
import numpy as np
import torch
import torch.optim as optim
from torchvision import datasets
from torch.utils.data import DataLoader
import albumentations as A
from albumentations.pytorch import ToTensorV2
def seed_everything(seed=42):
os.environ['PYTHONHASHSEED'] = str(seed)
np.random.seed(seed)
torch.manual_seed(seed)
torch.cuda.manual_seed(seed)
torch.cuda.manual_seed_all(seed)
torch.backends.cudnn.deterministic = True
torch.backends.cudnn.benchmark = False
class Transforms:
def __init__(self, img_size, mean_std, data='train'):
self.transforms = self.image_transform(img_size, mean_std, data)
def image_transform(self, img_size, mean_std, data):
if data == 'train':
return A.Compose(
[
A.Resize(img_size, img_size),
A.HorizontalFlip(p=0.5),
A.Normalize((mean_std[0], mean_std[1], mean_std[2]), (mean_std[3], mean_std[4], mean_std[5])),
ToTensorV2(),
]
)
elif data == 'val':
return A.Compose(
[
A.Resize(img_size, img_size),
A.Normalize((mean_std[0], mean_std[1], mean_std[2]), (mean_std[3], mean_std[4], mean_std[5])),
ToTensorV2(),
]
)
# (TTA)Test Time Augmentation
elif data == 'test':
return A.Compose(
[
A.Resize(img_size, img_size),
A.Normalize((mean_std[0], mean_std[1], mean_std[2]), (mean_std[3], mean_std[4], mean_std[5])),
ToTensorV2(),
]
)
def __call__(self, img, *args, **kwargs):
return self.transforms(image=np.array(img))['image']
class MetricMonitor:
def __init__(self, float_precision=3):
self.float_precision = float_precision
self.reset()
def reset(self):
self.metrics = defaultdict(lambda: {"val": 0, "count": 0, "avg": 0})
def update(self, metric_name, val):
metric = self.metrics[metric_name]
metric["val"] += val
metric["count"] += 1
metric["avg"] = metric["val"] / metric["count"]
def __str__(self):
return " | ".join(
[
"{metric_name}: {avg:.{float_precision}f}".format(
metric_name=metric_name, avg=metric["avg"],
float_precision=self.float_precision
)
for (metric_name, metric) in self.metrics.items()
]
)
def build_dataset(data_path, img_size, batch_size, mean_std):
train_dataset = datasets.ImageFolder(f'{data_path}/dataset/train', transform=Transforms(img_size, mean_std, 'train'))
val_dataset = datasets.ImageFolder(f'{data_path}/dataset/val', transform=Transforms(img_size, mean_std, 'val'))
test_dataset = datasets.ImageFolder(f'{data_path}/dataset/test', transform=Transforms(img_size, mean_std, 'test'))
train_loader = DataLoader(train_dataset, batch_size,
num_workers=0, pin_memory=True, shuffle=True, drop_last=False)
val_loader = DataLoader(val_dataset, batch_size,
num_workers=0, pin_memory=True, shuffle=False, drop_last=False)
test_loader = DataLoader(test_dataset, batch_size,
num_workers=0, pin_memory=True, shuffle=False, drop_last=False)
return train_loader, val_loader, test_loader
def build_optimizer(model, optimizer, learning_rate):
if optimizer == 'sgd':
optimizer = optim.SGD(model.parameters(),
lr=learning_rate, momentum=0.9)
elif optimizer == 'adam':
optimizer = optim.Adam(model.parameters(),
lr=learning_rate)
return optimizer
- util.py는 DataLoader, Image Transform 등과 같은 학습에 필요한 함수가 포함된 파일이다. 현재 데이터셋 구조가 라벨별로 데이터가 나뉘어 있기 때문에 Custom Dataset을 정의하지 않고 datasets.ImageFolder 모듈을 사용했다.
- 한가지 문제점이 num_workers=0 이 아닌 값을 주게 되면 shm(shared memory)가 OOM(Out Of Memory) 되었다고 에러가 발생한다. 이 문제는 도커에서도 발생하는 문제로 docker run에서 –shm-size를 지원하기 때문에 쉽게 해결되지만 쿠버네티스에서는 shm size를 위한 별도의 옵션을 지원하지 않는다. 쿠버네티스의 memory타입 emptyDir 마운트 방식으로 yaml 파일에서 직접 수정하는 방법이 있지만 현재 workspace-surface PVC를 통해 데이터셋을 가져와야 하는 상태라서 이 방법을 사용하지 못하는 상황이다.(추후 방법이 생기면 업데이트 할 예정)
- util.py는 hyperparameter-tuning, train, test 폴더에 각각 존재
DockerFile
FROM pytorch/pytorch:latest
RUN pip install -U numpy albumentations wandb tqdm timm
RUN mkdir -p /app
ARG DISABLE_CACHE
ADD . /app/
WORKDIR /app
ENTRYPOINT ["python3", "hyperparameter-wandb.py"]
component3: train-model
train.py
import argparse
import os
from tqdm import tqdm
import torch
import torch.nn as nn
import timm
from torch.optim.lr_scheduler import CosineAnnealingLR
from mlflow.pytorch import save_model
from mlflow.tracking.client import MlflowClient
from util import seed_everything, MetricMonitor, build_dataset, build_optimizer
class ConvNeXt(nn.Module):
def __init__(self, num_classes, pretrained=True):
super(ConvNeXt, self).__init__()
self.model = timm.create_model('convnext_tiny', pretrained=pretrained) # timm 라이브러리에서 pretrained model 가져옴
self.model.head.fc = nn.Linear(self.model.head.fc.in_features, num_classes, bias=True)
def forward(self, x):
return self.model(x)
def train_epoch(train_loader, epoch, model, optimizer, criterion, device):
metric_monitor = MetricMonitor()
model.train()
stream = tqdm(train_loader)
for i, (images, targets) in enumerate(stream, start=1):
images, targets = images.float().to(device), targets.to(device)
output = model(images)
loss = criterion(output, targets)
optimizer.zero_grad()
loss.backward()
optimizer.step()
predicted = torch.argmax(output, dim=1)
accuracy = round((targets == predicted).sum().item() / targets.shape[0] * 100, 2)
metric_monitor.update('Loss', loss.item())
metric_monitor.update('accuracy', accuracy)
stream.set_description(
f"Epoch: {epoch}. Train. {metric_monitor}"
)
def val_epoch(val_loader, epoch, model, criterion, device):
metric_monitor = MetricMonitor()
model.eval()
stream = tqdm(val_loader)
with torch.no_grad():
for i, (images, targets) in enumerate(stream, start=1):
images, targets = images.float().to(device), targets.to(device)
output = model(images)
loss = criterion(output, targets)
predicted = torch.argmax(output, dim=1)
accuracy = round((targets == predicted).sum().item() / targets.shape[0] * 100, 2)
metric_monitor.update('Loss', loss.item())
metric_monitor.update('accuracy', accuracy)
stream.set_description(
f"Epoch: {epoch}. Validation. {metric_monitor}"
)
return accuracy
def main(opt, device):
batch_size = 64
optimizer = 'sgd'
learning_rate = 0.001
epochs = 1
# read mean std values
with open(f'{opt.data_path}/mean-std.txt', 'r') as f:
cc = f.readlines()
mean_std = list(map(lambda x: x.strip('\n'), cc))
model = ConvNeXt(num_classes=2, pretrained=True)
model.to(device)
train_loader, val_loader, _ = build_dataset(opt.data_path, opt.img_size, batch_size, mean_std)
optimizer = build_optimizer(model, optimizer, learning_rate)
criterion = nn.CrossEntropyLoss()
scheduler = CosineAnnealingLR(optimizer, T_max=10,
eta_min=1e-6,
last_epoch=-1)
best_accuracy = 0
for epoch in range(1, epochs + 1):
train_epoch(train_loader, epoch, model, optimizer, criterion, device)
accuracy = val_epoch(val_loader, epoch, model, criterion, device)
scheduler.step()
if accuracy > best_accuracy:
os.makedirs(f'{opt.data_path}/weight', exist_ok=True)
torch.save(model.state_dict(), f'{opt.data_path}/weight/best.pth')
best_accuracy = accuracy
def upload_model_to_mlflow(opt, device):
os.environ["MLFLOW_S3_ENDPOINT_URL"] = "http://minio-service.kubeflow.svc:9000"
os.environ["AWS_ACCESS_KEY_ID"] = "minio"
os.environ["AWS_SECRET_ACCESS_KEY"] = "minio123"
client = MlflowClient("http://mlflow-server-service.mlflow-system.svc:5000")
model = ConvNeXt(num_classes=2, pretrained=False)
model.load_state_dict(torch.load(f'{opt.data_path}/weight/best.pth', map_location=device))
conda_env = {'name': 'mlflow-env', 'channels': ['conda-forge'],
'dependencies': ['python=3.9.4', 'pip', {'pip': ['mlflow', 'torch==1.8.0', 'cloudpickle==2.0.0']}]}
save_model(
pytorch_model=model,
path=opt.model_name,
conda_env=conda_env,
)
tags = {"DeepLearning": "surface crack classification"}
run = client.create_run(experiment_id="2", tags=tags)
client.log_artifact(run.info.run_id, opt.model_name)
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--data-path', type=str, help='dataset root path')
parser.add_argument('--img-size', type=int, help='resize img size')
parser.add_argument('--model-name', type=str, help='model name for artifact path')
parser.add_argument('--device', type=str, help='cuda device, i.e. 0 or 0,1,2,3 or cpu')
opt = parser.parse_args()
if opt.device == 'cpu':
device = 'cpu'
else:
os.environ["CUDA_VISIBLE_DEVICES"] = opt.device
device = "cuda" if torch.cuda.is_available() else "cpu"
print(f"DEVICE is {device}")
seed_everything()
main(opt, device)
upload_model_to_mlflow(opt, device)
- 하이퍼파라미터 값은 임의로 지정하였음
- upload_mode_to_mlflow 함수에서 학습된 모델을 mlflow experiment에 올리게 된다.
- minio는 Amazon S3와 호환하는 오브젝트 스토리지를 제공하는 오픈소스 소프트웨어이다. (Mlflow Tracking Server가 Artifacts Store로 사용할 용도로 쓰임)
- minio와 mlflow의 url이 IP 형태가 아닌 도메인 네임의 형태를 띄는것은 minio와 mlflow가 쿠버네티스에 배포되어있기 때문에, service name으로 dns lookup을 수행할 수 있기 때문이다.
- 129번 라인에서 create_run의 파라미터인 experiment_id는 mlflow의 experiment 번호를 의미하며 이는 mlflow ui에서 확인 가능하다. (아래 그림 참고)
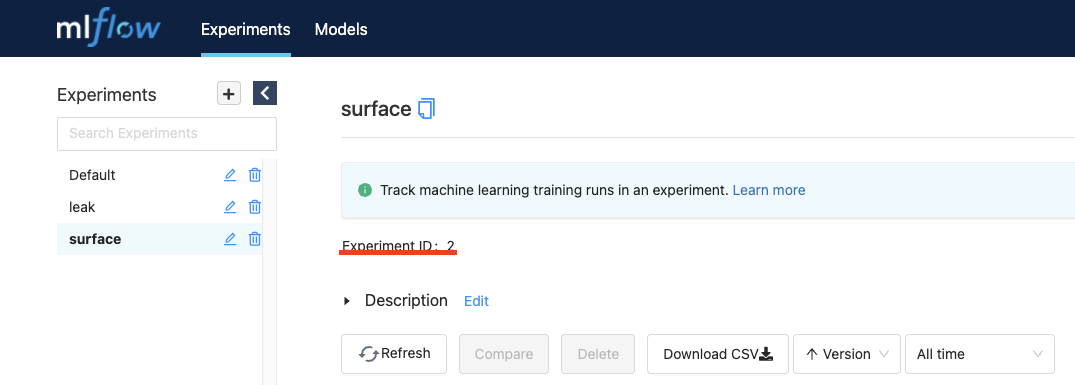
Dockerfile
FROM pytorch/pytorch:latest
RUN pip install -U numpy albumentations tqdm timm mlflow boto3
RUN mkdir -p /app
ARG DISABLE_CACHE
ADD . /app/
WORKDIR /app
ENTRYPOINT ["python", "train.py" ]
component4: test-model
test.py
import argparse
import os
from tqdm import tqdm
import torch
import torch.nn as nn
import mlflow
from util import seed_everything, MetricMonitor, build_dataset
def test(test_loader, model, criterion, device):
metric_monitor = MetricMonitor()
model.eval()
stream = tqdm(test_loader)
with torch.no_grad():
for i, (images, targets) in enumerate(stream, start=1):
images, targets = images.float().to(device), targets.to(device)
output = model(images)
loss = criterion(output, targets)
predicted = torch.argmax(output, dim=1)
accuracy = round((targets == predicted).sum().item() / targets.shape[0] * 100, 2)
metric_monitor.update('Loss', loss.item())
metric_monitor.update('accuracy', accuracy)
stream.set_description(
f"Test. {metric_monitor}"
)
def main(opt, device):
mlflow.set_tracking_uri("http://mlflow-server-service.mlflow-system.svc:5000")
os.environ["MLFLOW_S3_ENDPOINT_URL"] = "http://minio-service.kubeflow.svc:9000"
os.environ["AWS_ACCESS_KEY_ID"] = "minio"
os.environ["AWS_SECRET_ACCESS_KEY"] = "minio123"
batch_size = 64
with open(f'{opt.data_path}/mean-std.txt', 'r') as f:
cc = f.readlines()
mean_std = list(map(lambda x: x.strip('\n'), cc))
model = mlflow.pytorch.load_model(opt.model_path)
model.to(device)
_, _, test_loader = build_dataset(opt.data_path, opt.img_size, batch_size, mean_std)
criterion = nn.CrossEntropyLoss()
test(test_loader, model, criterion, device) # 마지막 iteration의 값들
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--data-path', type=str, help='dataset root path')
parser.add_argument('--img-size', type=int, help='resize img size')
parser.add_argument('--model-path', type=str, help='model path in mlflow, i,e. s3://~')
parser.add_argument('--device', type=str, help='cuda device, i.e. 0 or 0,1,2,3 or cpu')
opt = parser.parse_args()
if opt.device == 'cpu':
device = 'cpu'
else:
os.environ["CUDA_VISIBLE_DEVICES"] = opt.device
device = "cuda" if torch.cuda.is_available() else "cpu"
print(f"DEVICE is {device}")
seed_everything()
main(opt, device)
- mlflow의 모델을 불러와서 test를 진행하며 모델을 컨테이너에 저장하지는 않는다.
Dockerfile
FROM pytorch/pytorch:latest
RUN pip install -U numpy albumentations tqdm mlflow boto3 timm
RUN mkdir -p /app
ARG DISABLE_CACHE
ADD . /app/
WORKDIR /app
ENTRYPOINT ["python", "test.py" ]
각 컴포넌트를 하나씩 docker build & push를 하면 아래와 같이 도커허브에 이미지가 등록된다.
kfp pipeline
pipeline.py
import kfp
from kfp import dsl
from kfp import onprem
def preprocess_op(pvc_name, volume_name, volume_mount_path):
return dsl.ContainerOp(
name='Preprocess Data',
image='tjems6498/kfp-surface-preprocess:v0.1',
arguments=['--data-path', volume_mount_path,
'--img-size', 224],
).apply(onprem.mount_pvc(pvc_name, volume_name=volume_name, volume_mount_path=volume_mount_path))
def hyp_op(pvc_name, volume_name, volume_mount_path, device):
return dsl.ContainerOp(
name='Hyperparameter Tuning',
image='tjems6498/kfp-surface-hyp-wandb:v0.1',
arguments=['--data-path', volume_mount_path,
'--device', device],
).apply(onprem.mount_pvc(pvc_name, volume_name=volume_name, volume_mount_path=volume_mount_path))
def train_op(pvc_name, volume_name, volume_mount_path, device):
return dsl.ContainerOp(
name='Train Model',
image='tjems6498/kfp-surface-train:v0.1',
arguments=['--data-path', volume_mount_path,
'--img-size', 224,
'--model-name', 'surface-ConvNeXt-T',
'--device', device]
).apply(onprem.mount_pvc(pvc_name, volume_name=volume_name, volume_mount_path=volume_mount_path)).set_gpu_limit(4)
def test_op(pvc_name, volume_name, volume_mount_path, model_path, device):
return dsl.ContainerOp(
name='Test Model',
image='tjems6498/kfp-surface-test:v0.1',
arguments=['--data-path', volume_mount_path,
'--img-size', 224,
'--model-path', model_path,
'--device', device]
).apply(onprem.mount_pvc(pvc_name, volume_name=volume_name, volume_mount_path=volume_mount_path)).set_gpu_limit(4)
@dsl.pipeline(
name='Surface Crack Pipeline',
description=''
)
def surface_pipeline(mode_hyp_train_test: str,
preprocess_yes_no: str,
model_path: str,
device: str):
pvc_name = "workspace-surface"
volume_name = 'pipeline'
volume_mount_path = '/home/jovyan'
with dsl.Condition(preprocess_yes_no == 'yes'):
_preprocess_op = preprocess_op(pvc_name, volume_name, volume_mount_path)
with dsl.Condition(mode_hyp_train_test == 'hyp'):
_hyp_op = hyp_op(pvc_name, volume_name, volume_mount_path, device).after(_preprocess_op)
with dsl.Condition(mode_hyp_train_test == 'train'):
_train_op = train_op(pvc_name, volume_name, volume_mount_path, device).after(_preprocess_op)
with dsl.Condition(mode_hyp_train_test == 'test'):
_train_op = test_op(pvc_name, volume_name, volume_mount_path, model_path, device).after(_preprocess_op)
if __name__ == '__main__':
kfp.compiler.Compiler().compile(surface_pipeline, './surface.yaml')
- 모든 컴포넌트에서 데이터셋이 필요하기 때문에 onprem.mount_pvc 모듈을 통해 workspace-surface PVC를 mount 시켜준다.
- 주피터랩으로 PVC를 생성한 경우 volume root가 /home/jovyan으로 설정된다.
- surface_pipeline 함수의 파라미터는 kubeflow dashboard에서 사용자의 입력을 받는 용도로 쓰인다.
- dsl.Condition 모듈을 통해 DAG를 설계할 수 있다. 지금같은 경우에는 전처리가 필요한 경우 preprocess component를 실행하며 필요하지 않은 경우 바로 hyperparameter tuning, train, test중 한가지를 선택할 수 있게 하였다. (글 초반 DAG 그림 참고)
- 해당 파이썬 파일을 실행하면 쿠버네티스 manifest형식으로 surface.yaml 파일이 생성된다.
Kubeflow dashboard에 Pipeline 등록
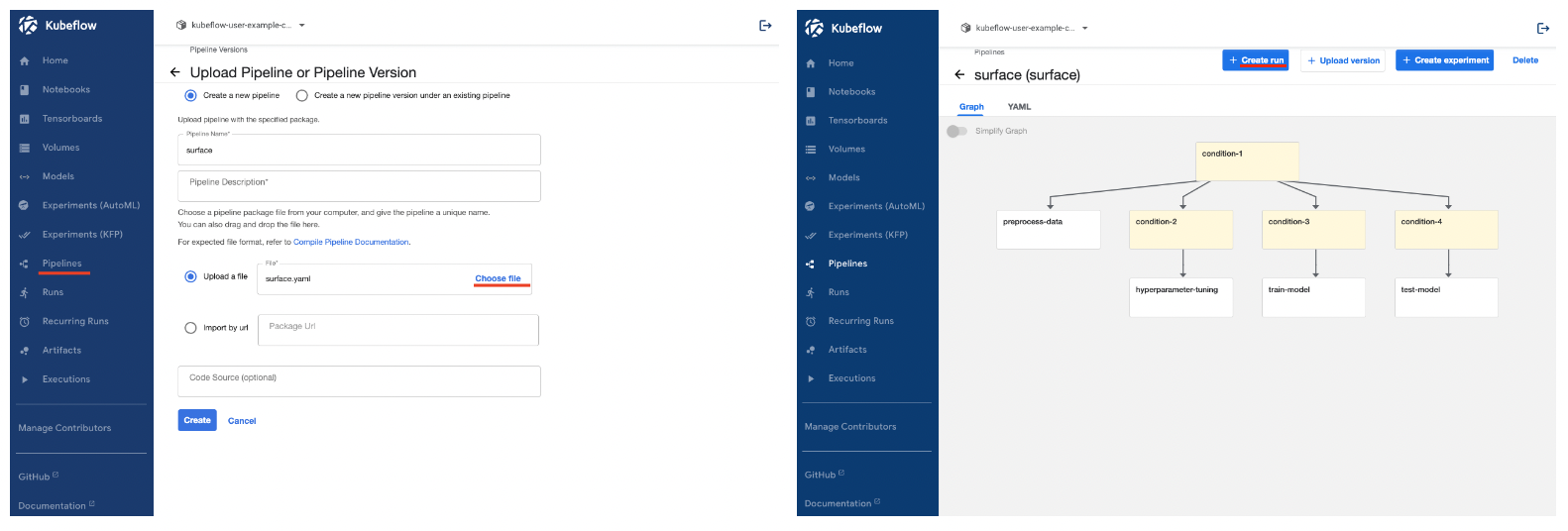
- 로컬에 있는 surface.yaml파일을 kubeflow dashboard pipeline 메뉴에서 파일을 업로드하여 파이프라인을 생성할 수 있다.
- pipeline을 생성하면 우측 그림처럼 DAG가 보이게 되고 create run 버튼으로 pipeline을 실행할 수 있다.
Experiments 생성
pipeline을 실행하기 위해 Experiments가 필요하다. Experiments는 단순히 프로젝트를 구분하는 용도로 쓰인다고 생각하면 된다.
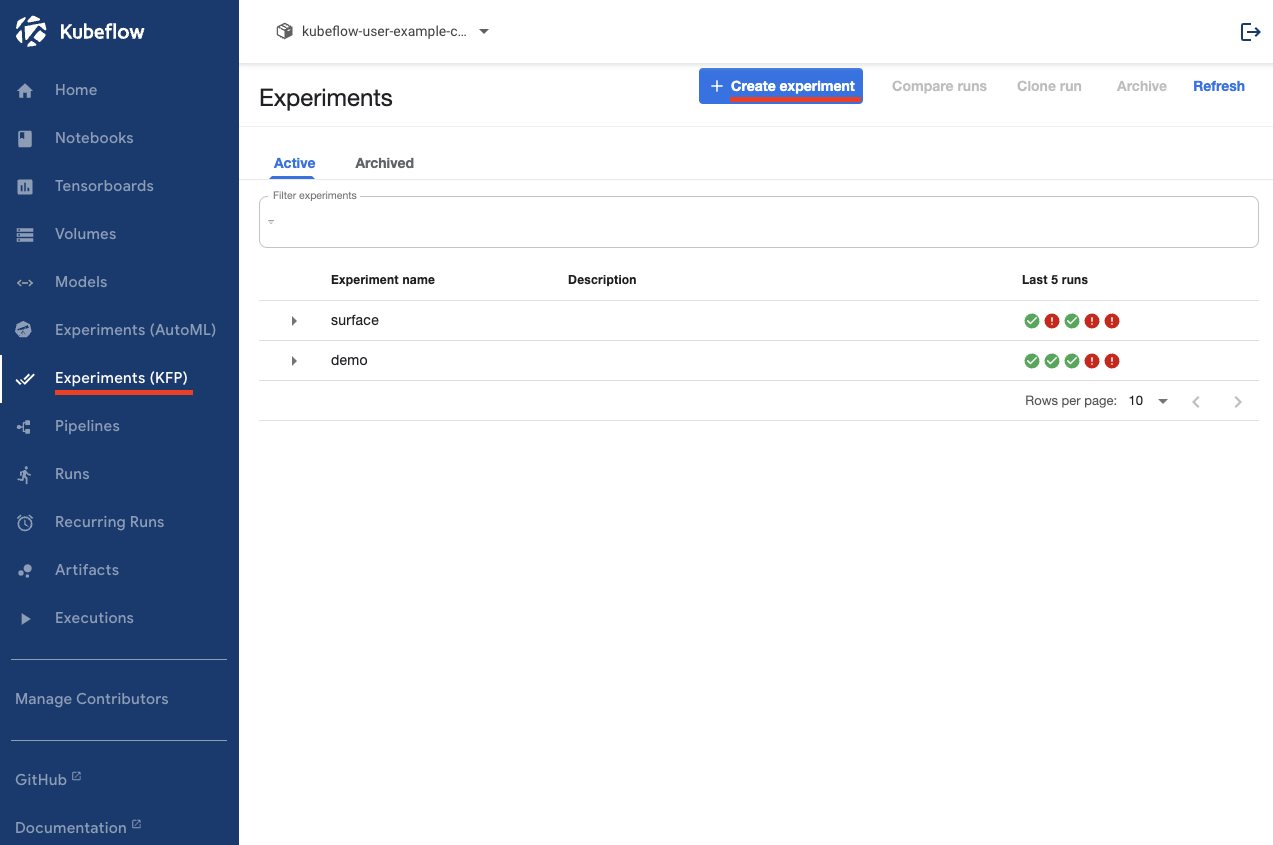
이제 Kubeflow pipeline을 실행할 준비가 되었다. 다음포스팅에 이어서 각 컴포넌트에 대해 어떤 결과가 나오는지 확인해보자

Leave a comment